VEP使用简介
- 1.背景知识
- 2.vep功能
-
- 2.1 可注释变异类型
- 2.2 注释结果信息
- 2.3 过滤筛选
- 3.软件下载与安装
-
- 3.1 安装vep的方式
- 4.vep的使用
-
- 4.1.使用基本的参数进行测试
- 4.2 在docker下使用
1.背景知识
二代测序发现的变异数量是巨大的,以人类基因组为例,1个人类基因组包含近3500000个SNV突变和1000个拷贝数变异,其中约20000-25000个变异是在编码区,10000个位点发生了氨基酸编码改变,仅有50-100个发生了蛋白截断或功能缺失,手动检查每个位点非常耗时且有些不切实际。
Variant Effect Predictor(VEP)是一个功能强大的工具集,用于编码和非编码区域的基因组变异的分析、注释和优先排序。它提供了对广泛的基因组注释集合的访问,具有各种接口以适应不同的需求,以及用于配置和扩展分析的简单参数。它是开源的,免费使用,并支持结果完全复现。
默认情况下,VEP 使用基于归一化的等位基因匹配算法来识别与输入变体匹配的已知变体。由于输入和已知变体可能具有多个替代(ALT)或变体等位基因,因此每对参考(REF)和ALT等位基因都经过归一化并独立比较以达到潜在的匹配。VCF 允许在同一行上编码多个等位基因类型,而 dbSNP 将单独的 rsID 标识符分配给同一位点的不同等位基因类型。这意味着来自同一输入变体的不同等位基因可能会被分配不同的已知变体标识符。
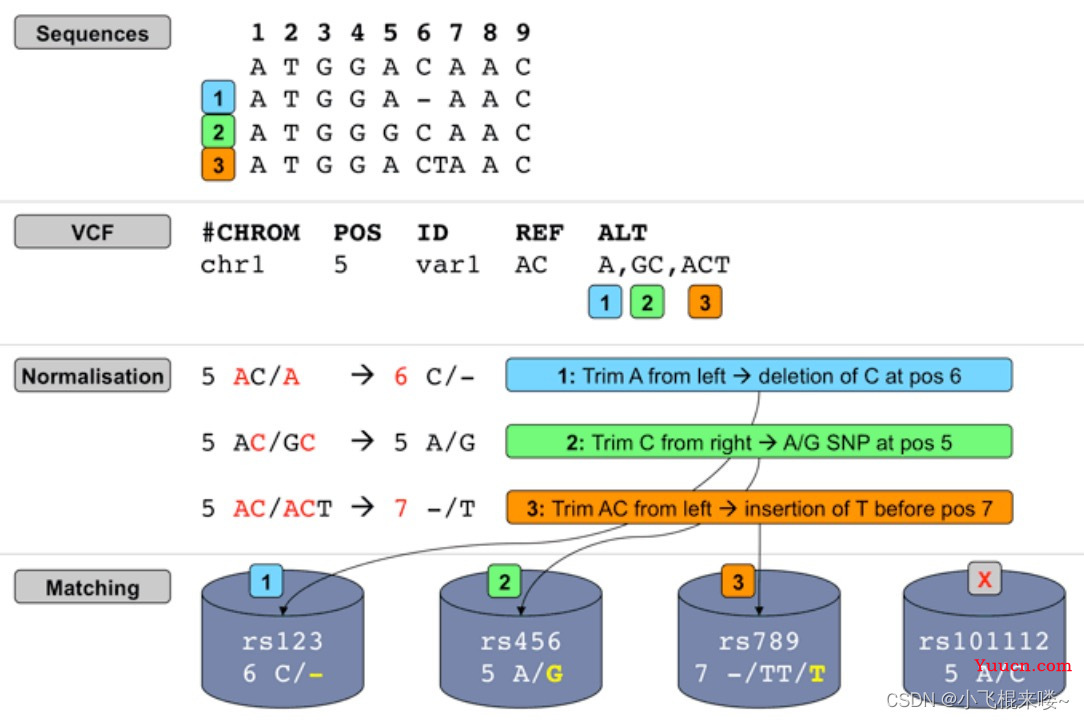
并且,VEP 支持使用 HGVS 作为输入。此功能目前正在开发中,并非所有 HGVS 表示法类型都受支持。完全支持与基因组 (g.) 或编码 (c.) 序列相关的符号;蛋白质(P.)符号以有限的方式得到支持,因为确定可能产生单个蛋白质变化的多种可能的潜在基因组序列变化涉及复杂性。如果无法解析特定表示法,将发出警告。
2.vep功能
2.1 可注释变异类型
明确位置及序列改变的变异,全外显子组和全基因组检测出的变异,包含SNVs,insertions、deletion等。
VEP支持多种物种,也可自行构建数据库,不过使用ensemble数据库中所注释好的内容为最佳选择,可在https://ftp.ensembl.org/pub/release-108/variation/ 查看是否支持该物种。
2.2 注释结果信息
(1)功能注释信息,主要是把变异在基因组的位置和功能影响标出。包括基因信息和变异影响,信息来自RefSeq或Ensembl;
(2)频率信息,标记群体数据库频率,包含数据库总的频率及子群体频率。这部分信息主要在疾病研究中根据疾病的流行病学研究,有针对性的筛选低频或罕见变异,为人类疾病研究提供基础。高通量测序检出的变异数目众多,对于遗传病来说,要筛选出致病变异,需要过滤掉一些常见变异,群体变异库的使用可以进一步缩小筛选范围;
(3)软件预测结果信息,突变的致病性预测。可有效缩小变异范围,协助研究者更快找到致病基因;
(4)疾病数据库信息,HGMD、Clinvar等数据库信息,协助用户快速定位已知致病位点;
2.3 过滤筛选
VEP提供注释结果的简单过滤,主要包含:
(1)筛选功能突变,筛选出coding区和剪切区域的突变;
(2)按照频率过滤,筛选出低于指定频率的突变;
(3) 按照遗传模式过滤。
3.软件下载与安装
3.1 安装vep的方式
(1)安装vep
a. 直接使用conda来安装vep软件,省去了多余复杂的配置问题,但是由于vep涉及到的软件版本和依赖较多,因此建议新建一个虚拟环境来安装
conda create -n vep # 需要创建一个虚拟环境安装vep
conda activate vep # 激活环境
conda install -c bioconda ensembl-vep==108 ## 安装vpe,108是最新版本
b. 使用docker安装
docker pull ensemblorg/ensembl-vep
(2)下载vep相应的数据库
cache 数据集的下载
cache是一个可下载的文件,包含一个物种的所有转录模型,调控特征和变异数据。下载使用cache是使用VEP的最快和最有效的方法,因为在大多数情况下,只建立一个初始网络连接,大多数数据是从本地磁盘读取的。使用离线模式消除所有网络连接的速度和/或隐私。cache 只有两种类型,分别是refseq和merged。在此基础上,VEP运行时还可以使用plugin和custom参数自定义添加数据库。
注意:强烈建议下载/使用对应于Ensembl VEP安装的VEPcache版本,即VEP cache版本108应该与Ensembl VEP工具版本108一起使用。这主要是由于VEP cache(数据内容和结构)是在每个Ensembl版本中生成的,关于此版本的数据和API更新,因此cache数据格式可能在版本之间有所不同(并且与新版本的Ensembl VEP工具不兼容)。
cache 包含的信息:转录本位置,序列,外显子和其他属性;每个转录本的基因、蛋白质、 HGNC 和其他标识符(在适用情况下,仅适用于 RefSeq cache);现有变异的位置、等位基因和频率;调控区域;SIFT,PolyPhen 的预测和分数;
cache下载方式:进入vep官网:https://ftp.ensembl.org/pub/release-108/variation/
点击vep 进入新的界面,选择需要的的物种和相应版本的cache文件包,点击右键,复制链接,在服务器中wget下载;
(3) 下载vep相关的插件
vep插件放在github公共仓库中(链接:https://github.com/Ensembl/VEP_plugins),根据需要下载相应的插件;
vep官网有vep插件的详细介绍(链接:https://asia.ensembl.org/info/docs/tools/vep/script/vep_plugins.html);
4.vep的使用
4.1.使用基本的参数进行测试
/opt/vep/src/ensembl-vep/vep -i ${sample}.vcf \ # 输入的vcf文件
--dir_cache ${dir_cache} \ # cache文件包路径
--fork 4 \ # 线程数
--refseq /--merged \ # cache文件包的种类,vep 的cache文件包只有两种,分别是refseq,merged
--format vcf \
--output_file test.vcf \
--custom ${dir_plugins}/MT.mitomap.vcf.gz,Mitomap,vcf,exact,0,AC,AF,Disease,DiseaseStatus \
--offline \
--assembly GRCh37 \
--use_given_ref \
--vcf \
--force_overwrite \
4.2 在docker下使用
docker run -t -i -v $HOME/vep_data:/opt/vep/.vep ensemblorg/ensembl-vep
vep官网中有关运行vep有很多参数,比如 Basic options 、Cache options 、Other annotation sources、 Output format options 等,其包含很多参数选项和简介,根据不同的需求选择合适的参数;vep参数的网站 https://asia.ensembl.org/info/docs/tools/vep/script/vep_options.html